Struttura a doppia elica del DNA
Il DNA ha una struttura a doppia elica (mostrata sotto) creata da due filamenti di subunità nucleotidiche legate covalentemente. I gruppi di zucchero e fosfato di ciascun filamento di nucleotidi sono posizionati all’esterno dell’elica, formando la spina dorsale del DNA (evidenziata dai nastri arancioni nella Figura 3). I due filamenti dell’elica corrono in direzioni opposte, il che significa che l’estremità 5′ del carbonio di un filamento è rivolta verso l’estremità 3′ del carbonio del filamento corrispondente (vedi figure 4 e 5). Ci siamo riferiti a questo orientamento dei due filamenti come antiparallelo. Si noti anche che i gruppi fosfato sono rappresentati nella Figura 3 come “bastoncini” arancioni e rossi che sporgono dal nastro. I fosfati sono caricati negativamente a pH fisiologici e quindi danno alla spina dorsale del DNA un forte carattere locale caricato negativamente. Al contrario, le basi azotate sono impilate all’interno dell’elica (queste sono rappresentate come bastoncini verdi, blu, rossi e bianchi nella Figura 3). Le coppie di nucleotidi interagiscono tra loro attraverso specifici legami idrogeno (mostrati nella Figura 5). Ogni coppia è separata dalla coppia di basi successiva nella scala di 0,34 nm e questo stretto impilamento e l’orientamento planare danno luogo a interazioni base-impilamento energeticamente favorevoli. La chimica specifica associata a queste interazioni va oltre il contenuto di Bis2a, ma è descritta più in dettaglio qui per i curiosi o gli studenti più avanzati. Ci aspettiamo, tuttavia, che gli studenti siano consapevoli del fatto che l’impilamento delle basi azotate contribuisce alla stabilità della doppia elica e rimandiamo ai vostri istruttori di genetica e chimica organica di divisione superiore per completare i dettagli chimici.
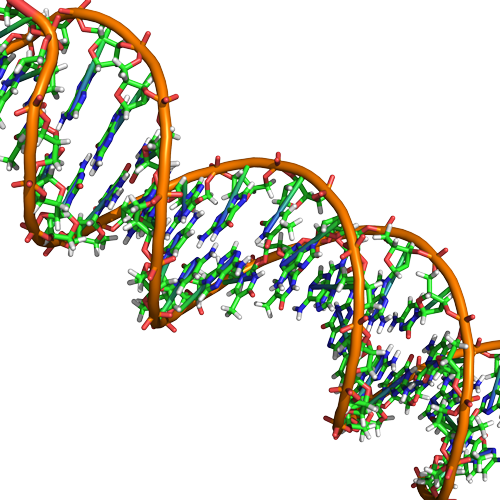
Figura 3. Il DNA nativo è una doppia elica antiparallela. La spina dorsale di fosfato (indicata dalle linee curve) è all’esterno, e le basi sono all’interno. Ogni base di un filamento interagisce tramite legame a idrogeno con una base del filamento opposto. Attribuzione: Marc T. Facciotti (lavoro originale)
In una doppia elica, certe combinazioni di accoppiamento delle basi sono chimicamente più favorite di altre in base ai tipi e alle posizioni dei gruppi funzionali sulle basi azotate di ogni nucleotide. In biologia troviamo che:
Adenina (A) è chimicamente complementare con timidina (T) (A si accoppia con T)
e
Guanina (G) è chimicamente complementare con citosina (C) (G si accoppia con C).
Spesso ci riferiamo a questo schema come “complementarietà di base” e diciamo che i filamenti antiparalleli sono complementari l’uno all’altro. Per esempio, se la sequenza di un filamento di DNA è 5′-AATTGGCC-3′, il filamento complementare avrebbe la sequenza 5′-GGCCAATT-3′.
A volte scegliamo di rappresentare le strutture complementari a doppia elica nel testo impilando i filamenti complementari uno sull’altro come segue:
5′ – GGCCAATTCCATACTAGGT – 3′
3′ – CCGGTTAAGGTATGATCCA – 5′
Nota che ogni filamento ha le sue estremità 5′ e 3′ etichettate e che se uno dovesse camminare lungo ogni filamento partendo dall’estremità 5′ verso l’estremità 3′ la direzione del viaggio sarebbe opposta all’altra per ogni filamento; i filamenti sono antiparalleli. Diciamo comunemente cose come “percorrere 5-prime a 3-prime” o “sintetizzare 5-prime a 3-prime” per riferirci alla direzione in cui stiamo leggendo una sequenza o alla direzione della sintesi. Cominciate ad abituarvi a questa nomenclatura.
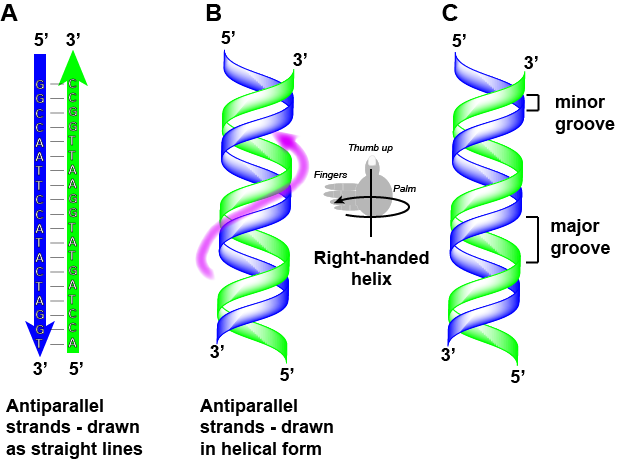
Attribuzione: Marc T. Facciotti (lavoro originale)
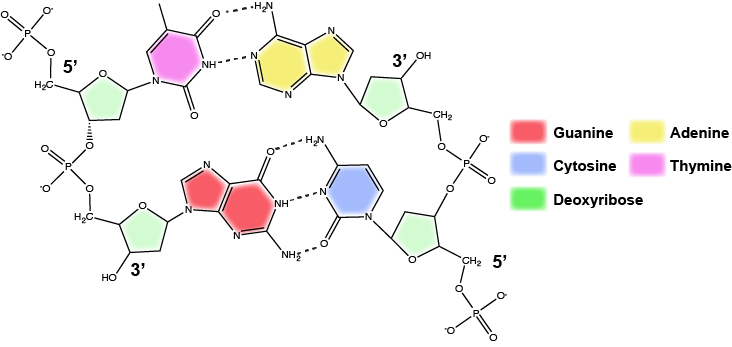
Attribuzione: Marc T. Facciotti (lavoro originale)