Doppelhelixstruktur der DNA
Die DNA hat eine Doppelhelixstruktur (siehe unten), die aus zwei Strängen kovalent verbundener Nukleotiduntereinheiten besteht. Die Zucker- und Phosphatgruppen jedes Nukleotidstrangs befinden sich auf der Außenseite der Helix und bilden das Rückgrat der DNA (hervorgehoben durch die orangefarbenen Bänder in Abbildung 3). Die beiden Stränge der Helix verlaufen in entgegengesetzter Richtung, was bedeutet, dass das 5′-Kohlenstoffende eines Strangs dem 3′-Kohlenstoffende seines Gegenstrangs gegenüberliegt (siehe Abbildungen 4 und 5). Wir bezeichnen diese Ausrichtung der beiden Stränge als antiparallel. Beachten Sie auch, dass die Phosphatgruppen in Abbildung 3 als orangefarbene und rote „Stäbchen“ dargestellt sind, die aus dem Band herausragen. Die Phosphate sind bei physiologischen pH-Werten negativ geladen und verleihen daher dem Rückgrat der DNA einen starken lokalen negativ geladenen Charakter. Im Gegensatz dazu sind die stickstoffhaltigen Basen im Inneren der Helix gestapelt (diese sind in Abbildung 3 als grüne, blaue, rote und weiße Stäbchen dargestellt). Paare von Nukleotiden interagieren miteinander durch spezifische Wasserstoffbrückenbindungen (dargestellt in Abbildung 5). Jedes Paar ist vom nächsten Basenpaar in der Leiter um 0,34 nm getrennt, und diese enge Stapelung und planare Ausrichtung führt zu energetisch günstigen Base-Stacking-Wechselwirkungen. Die spezifische Chemie, die mit diesen Wechselwirkungen verbunden ist, geht über den Inhalt von Bis2a hinaus, wird aber hier für die neugierigen oder fortgeschrittenen Schüler ausführlicher beschrieben. Wir gehen jedoch davon aus, dass die Studenten wissen, dass die Stapelung der stickstoffhaltigen Basen zur Stabilität der Doppelhelix beiträgt, und überlassen es den Dozenten für Genetik und organische Chemie, die chemischen Details zu erläutern.
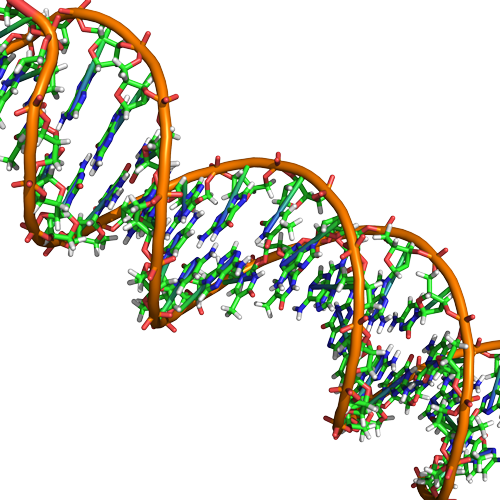
Abbildung 3. Native DNA ist eine antiparallele Doppelhelix. Das Phosphatgerüst (angedeutet durch die geschwungenen Linien) befindet sich auf der Außenseite und die Basen sind auf der Innenseite. Jede Base des einen Strangs interagiert über Wasserstoffbrückenbindungen mit einer Base des gegenüberliegenden Strangs. Zuschreibung: Marc T. Facciotti (Originalarbeit)
In einer Doppelhelix sind bestimmte Kombinationen von Basenpaarungen chemisch bevorzugter als andere, basierend auf den Arten und Positionen der funktionellen Gruppen auf den stickstoffhaltigen Basen jedes Nukleotids. In der Biologie finden wir, dass:
Adenin (A) ist chemisch komplementär zu Thymidin (T) (A paart sich mit T)
Guanin (G) ist chemisch komplementär zu Cytosin (C) (G paart sich mit C).
Wir bezeichnen dieses Muster oft als „Basenkomplementarität“ und sagen, dass die antiparallelen Stränge komplementär zueinander sind. Wenn zum Beispiel die Sequenz eines Strangs der DNA 5′-AATTGGCC-3′ ist, würde der komplementäre Strang die Sequenz 5′-GGGCCAATT-3′ haben.
Manchmal wählt man die Darstellung komplementärer Doppelhelix-Strukturen im Text, indem man die komplementären Stränge wie folgt übereinander stapelt:
5′ – GGCCAATTCCATACTAGGT – 3′
3′ – CCGGTTAAGGTATGATCCA – 5′
Beachten Sie, dass jeder Strang an seinem 5′- und 3′-Ende beschriftet ist und dass, wenn man an jedem Strang vom 5′-Ende zum 3′-Ende laufen würde, die Laufrichtung für jeden Strang entgegengesetzt wäre; die Stränge sind antiparallel. Wir sagen umgangssprachlich Dinge wie „5-prime zu 3-prime laufen“ oder „5-prime zu 3-prime synthetisieren“, um die Richtung zu bezeichnen, in der wir eine Sequenz lesen oder die Richtung der Synthese. Gewöhnen Sie sich an diese Nomenklatur.
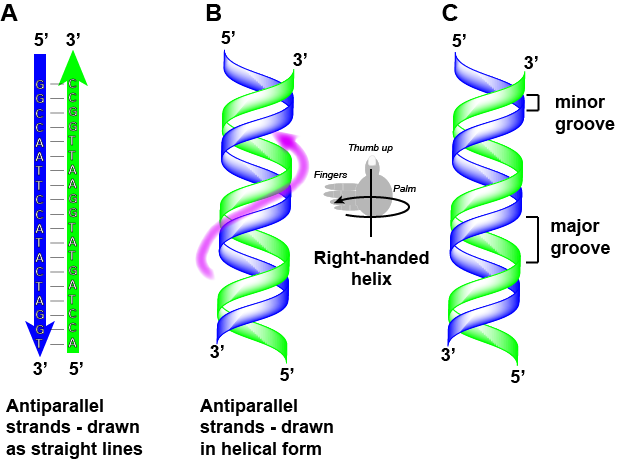
Attribution: Marc T. Facciotti (Originalarbeit)
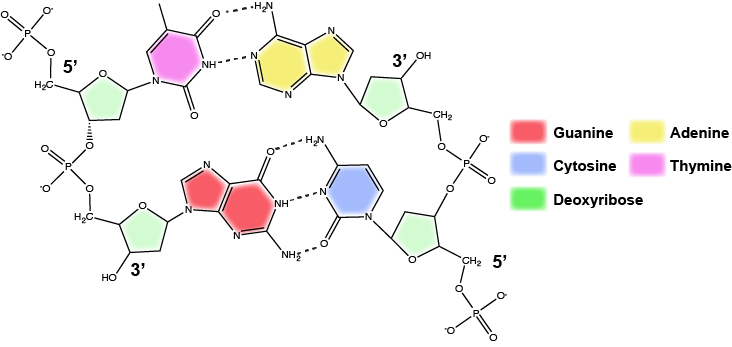
Attribution: Marc T. Facciotti (Originalarbeit)